近日,Advanced Science在线发表了江南大学未来食品科学中心陈坚院士团队刘龙教授课题组的研究成果“Reshaping Phosphatase Substrate Preference for Controlled Biosynthesis Using a “Design–Build–Test–Learn” Framework”(Lu et al.,Adv.Sci., 2024, 2309852)。2020级博士生卢建功为论文第一作者,刘龙教授为通讯作者。
酶在微生物细胞工厂中扮演着重要角色。然而,自然进化而来的野生型酶存在催化性能不足的问题,通常需要利用蛋白质工程改造来满足工业生产的需求。在缺乏蛋白质结构和催化机制等信息的情况下,基于定向进化的非理性策略可以获得正向突变,但庞大的突变体文库规模严重限制了改造效率。目前,众多蛋白质三维结构和催化机制的逐步解析使半理性策略作为一种替代方法,通过识别影响酶性能的关键位点显著缩小了突变体文库的规模。然而,这种半理性策略对于多位点迭代突变仍存在效率较低的问题。
针对上述问题,该研究集成了分子动力学模拟、量子力学计算以及结构生物学等多学科研究策略,提出了一个设计-构建-测试-学习(DBTL)迭代框架,通过理性设计显著改善了磷酸酶BT4131的催化活性及底物专一性,并大大促进了N-乙酰氨基葡萄糖(GlcNAc)微生物细胞工厂的生产效率。
来自Bacteroides thetaiotaomicronVPI-5482的磷酸酶BT4131对GlcNAc的前体N-乙酰氨基葡萄糖-6-磷酸(GlcNAc6P)和葡萄糖-6-磷酸(Glc6P)均具有较强的催化活性,这限制了其在GlcNAc生物合成中的应用。该研究首先通过分子动力学模拟确定了影响底物偏好性的关键结合位点,并计算了这些位点的氨基酸残基突变对不同底物亲和力的影响。然后,根据计算结果构建并测试了突变体L129Q,其kcat-GlcNAc6P/Km-GlcNAc6P增加了1.4倍,kcat-Glc6P/Km-Glc6P减少了73.1%。
随后利用基于密度泛函(DFT)的量子力学(QM)计算进行了催化反应过程的势能面扫描,并针对高能过渡态构象预测了迭代突变体-底物亲和力。进而通过增强亲和力稳定过渡态构象来降低反应能垒,由此获得了催化效率进一步改善的突变体M4(I49Q/L129Q/G172L),其kcat-GlcNAc6P/Km-GlcNAc6P值比野生型增加了9.5倍,kcat-Glc6P/Km-Glc6P值减少了59%。
为了使磷酸酶表达强度与胞内GlcNAc6P积累程度相耦连来进一步促进GlcNAc的合成,利用来自大肠杆菌的nagC转录因子构建了GlcNAc6P响应型生物传感器,并通过将nagC结合序列插入到启动子的-35和-10等不同区域优化了传感器的响应阈值及灵敏度。此外,进一步结合了乳糖操纵子元件使传感器同时受到GlcNAc6P以及IPTG的双重调控。
最终,将突变体M4与上述生物传感器结合并应用于高产GlcNAc的大肠杆菌中,在50升生物反应器中GlcNAc产量达到了217.3 g L-1,提高了25.8%,转化率为0.597 g g-1葡萄糖,均为目前报道的最高水平。因此,该研究获得了一种对GlcNAc6P具有显著偏好性的磷酸酶突变体,也为其他酶的蛋白质工程理性设计提供了有价值的工具。
本研究得到国家重点研发计划(2020YFA0908300)的资助。

图1 磷酸酶底物偏好性改造的DBTL框架
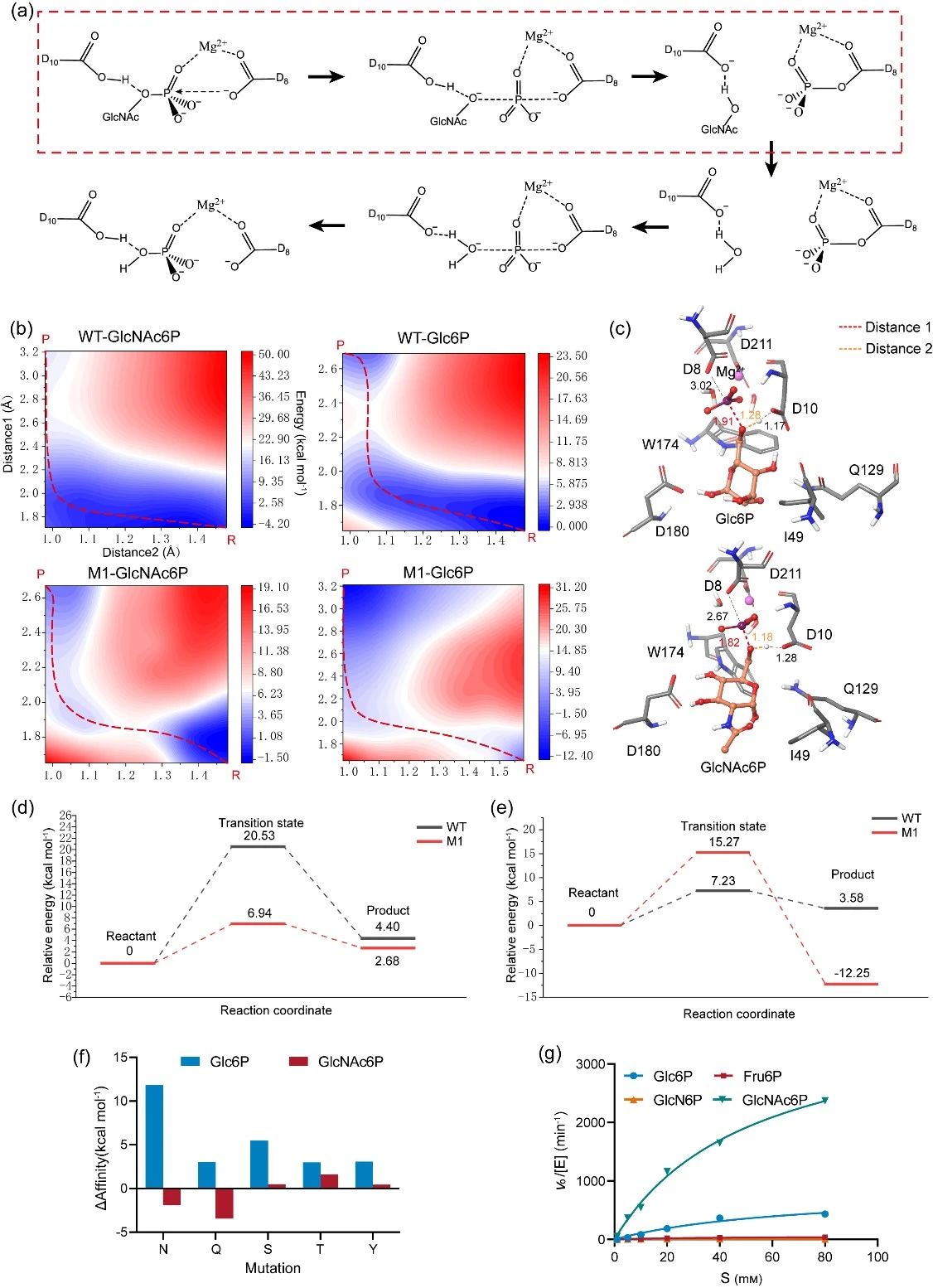
图2 催化反应过程的量子力学计算结果及迭代突变
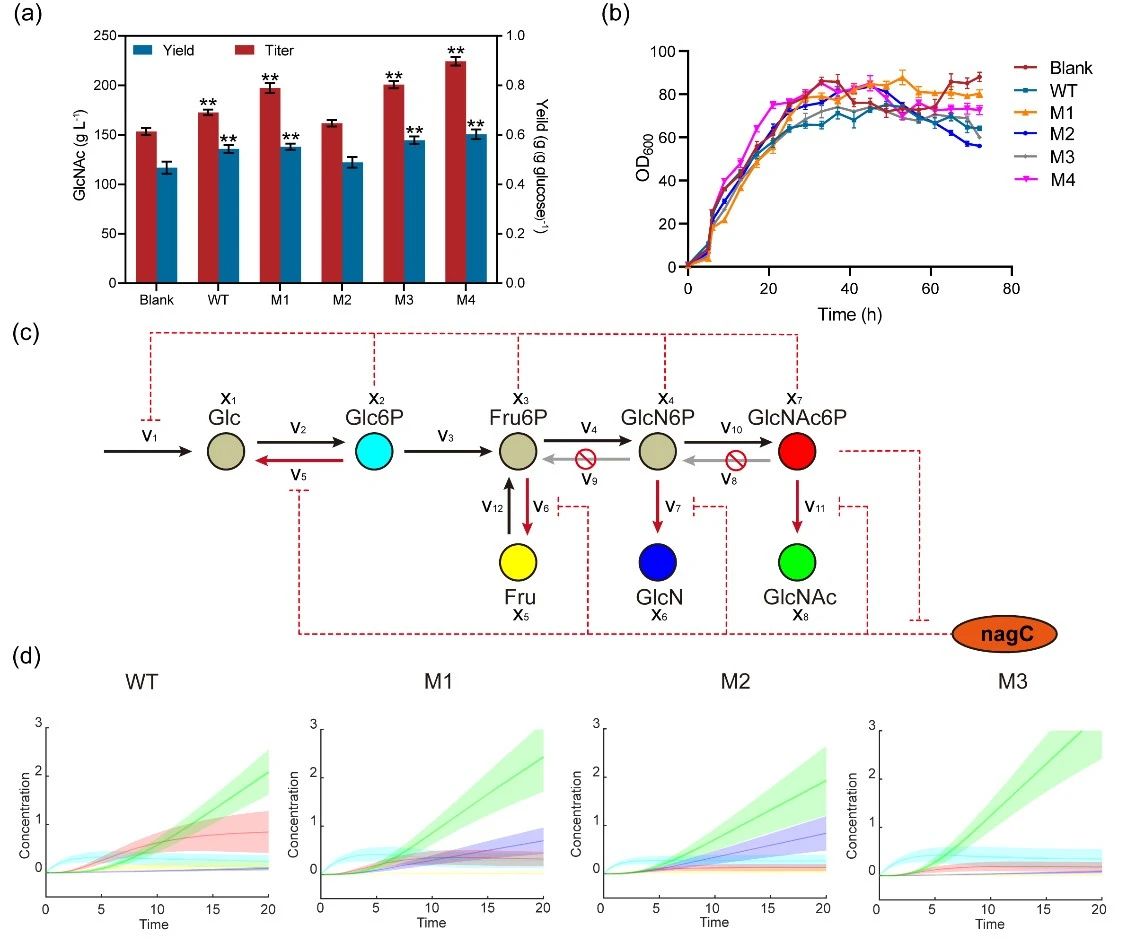
图3 磷酸酶突变体在GlcNAc生物合成中的应用及动力学模型分析
(编辑:潘梦妍)
